Umgang mit “Missing values”
In den Bemühungen um möglichst hohe Datenqualität geht es heute um eine bestimmte Form von Datenbank-Auffälligkeit - die fehlenden Werte oder Missing Values/Missings. Fehlende Werte können zufällig über die Datenbank gestreut sein - da es unvermeidlich ist, in einer großen Datenfülle nicht immer alle Werte erheben zu können. Trotzdem wäre es wichtig, solche fehlenden Werte zu identifizieren, und in der anstehenden statistischen Analyse zu versuchen, zumindest einige Missings durch Ausnützen von Redundanzen in der Datenbank durch “richtige” Werte zu ersetzen.
Noch wichtiger ist es, systematisch als fehlend auftretende Werte zu identifizieren. Dies sollte so früh wie möglich im Ablauf der Datenerfassung passieren, um bei der Datenerfassung bestehende Probleme aufdecken und die Datenerfassung möglichst schnell komplettieren zu können.
Wir sind also jetzt beim zweiten Schritt der Datenbereinigung:
Wie schon angedeutet, ist R eine ausgesprochen mächtige, und (als freie Variante der Statistik-Sprache S) für statistische Auswertungen besonders gut geeignete Programmiersprache. Zu den Charakteristika dieser freien 1 Sprache gehört die Verfügbarkeit vieler Funktionen für besondere Zwecke, die auch weit über den ursprünglichen Rahmen hinausgehen. Diese Funktionen werden im Rahmen von Bibliotheks-“Paketen” bei Bedarf in die Software eingebunden. Auch dieser Blog wird mit Hilfe von R-Paketen erstellt!
Das nachfolgende Code-Fragment, das wir bereits als Beispiel für eine R-Erweiterung vorgestelt hatten, bindet zusätzlich die Bibliothek “overviewR” 2 ein, die für die einfache Darstellung von Daten konzipiert wurde.
library(tidyverse)
library(overviewR)
GTDS_NA <- overviewR::overview_na(GTDSroh)Tatsächlich wollen wir uns, für einen ersten Überblick, nur einige Datenbank-Parameter anschauen. WIr wählen deshalb unter den Tabellen-Spalten der in einem früheren Beitrag erstellten Datei “GTDSroh” einige aus, bevor die Darstellung des Anteils von fehlenden Werten ausgelöst wird. Dies gelingt gut mit der Funktion “select” aus der Bibliothek “dplyr” des “Tidyverse”. Hierbei ist die Namensgebung der einzelnen Variablen (Spalten) unerheblich, und entspricht in diesem Fall der Konvention von exportierten GTDS-Daten:
library(tidyverse)
library(overviewR)
GTDS_select <- GTDSroh %>% dplyr::select(SEX,DIAALTER,DIA_DAT,DIAICD10,HIST_DAT,OP_DAT,STERBDAT)
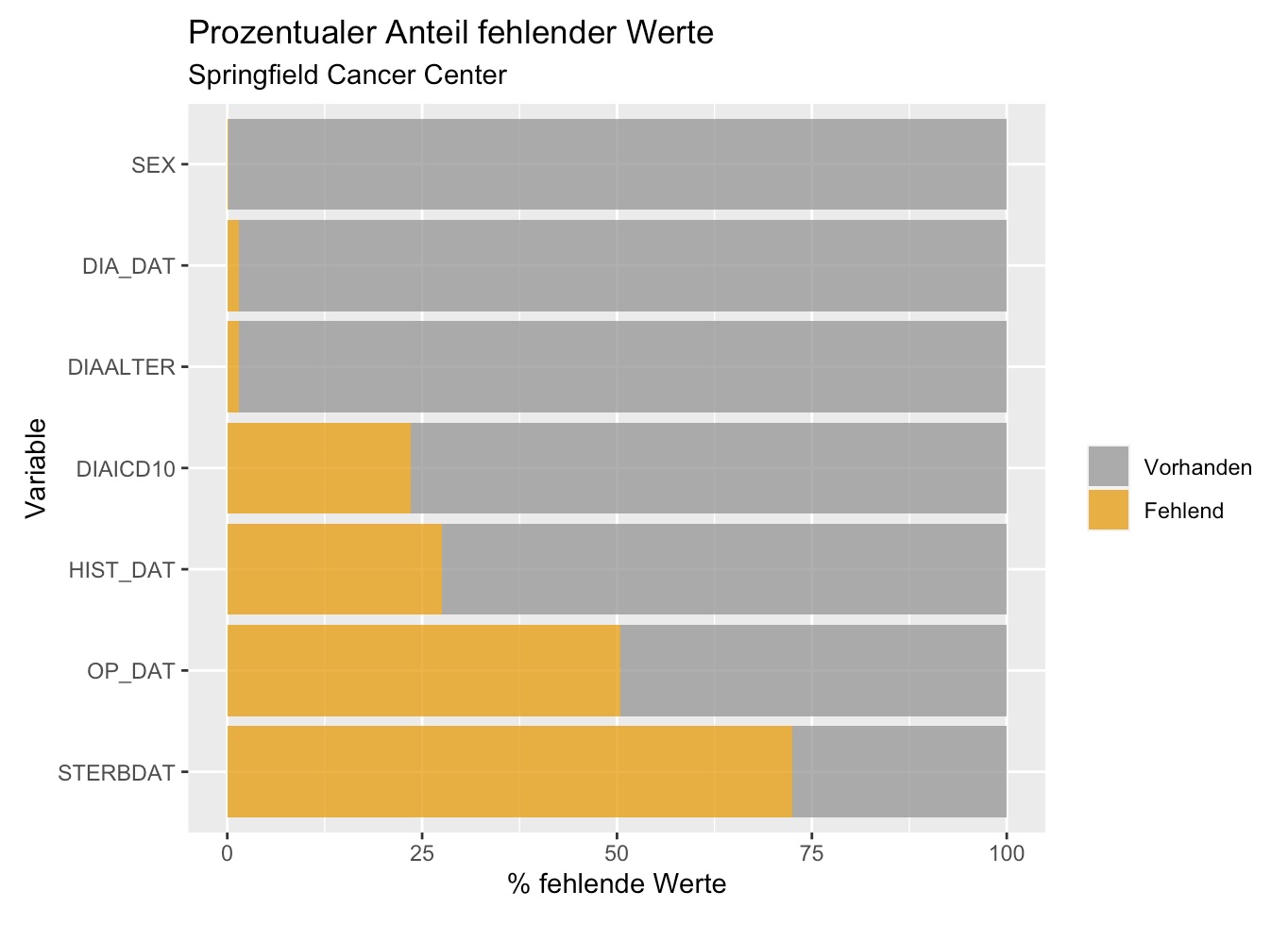
GTDS_NA <- overviewR::overview_na(GTDS_select)Das Ergebnis ist ein Barplot, aus dem der prozentuale Anteil fehlender Werte eines Datenbank-Parameters abgelesen werden kann.
library(DataExplorer)
Attaching package: 'DataExplorer'The following object is masked from 'package:expss':
split_columnsmiss_plot <- plot_missing(GTDS_select)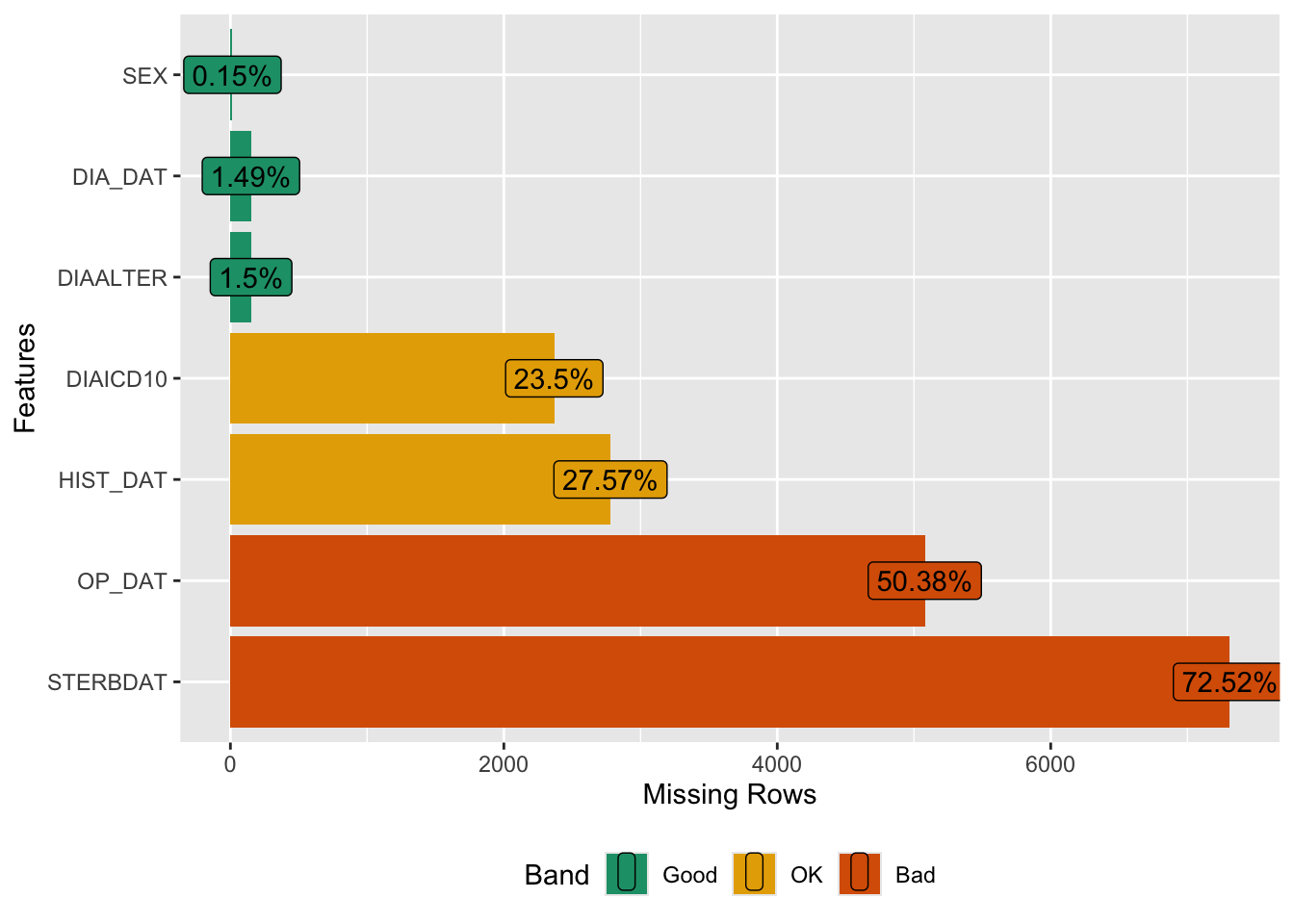
Man kann sehen, dass bei der Dateneingabe das Patientengeschlecht (SEX) praktisch immer vorlag, und sowohl das Diagnosedatum (DIA_DAT) als auch Alter des Patienten bei Diagnosestellung (DIAALTER) nur ganz selten fehlt. Im Kontrast dazu steht das Sterbedatum, das bei den meisten Fällen fehlt. Dies ist natürlich sehr verständlich, weil die Überlebensrate nach Krebs heutzutage sehr hoch ist, und deshalb bei den meisten Fällen ein Sterbedatum nicht angegeben werden kann.
Man sieht auch, dass nur etwa die Hälfte der in der Datenbank erfassten Patienten in der primären Behandlungsphase operiert wurde, und bei manchen Patienten das Datum der histologischen Erstdiagnose fehlt - möglicherweise Patienten, die erst im Erkrankungsverlauf zum Zentrum gestoßen sind, und die Primärdaten womöglich nicht mehr erreichbar waren. Dieses Zentrum ist allerdings stark in der Behandlung von Leukämien. Leukämien werden in der Regel aus dem Blut, und nicht histologisch gesichert.
Vor Ort müsste letztlich entschieden werden, ob eventuell doch eine systematische Untererfassung bestimmter Daten vorliegen könnte - man wird sich z.B. den Prozess der Erfassung der histologischen Befunde genauer ansehen müssen.
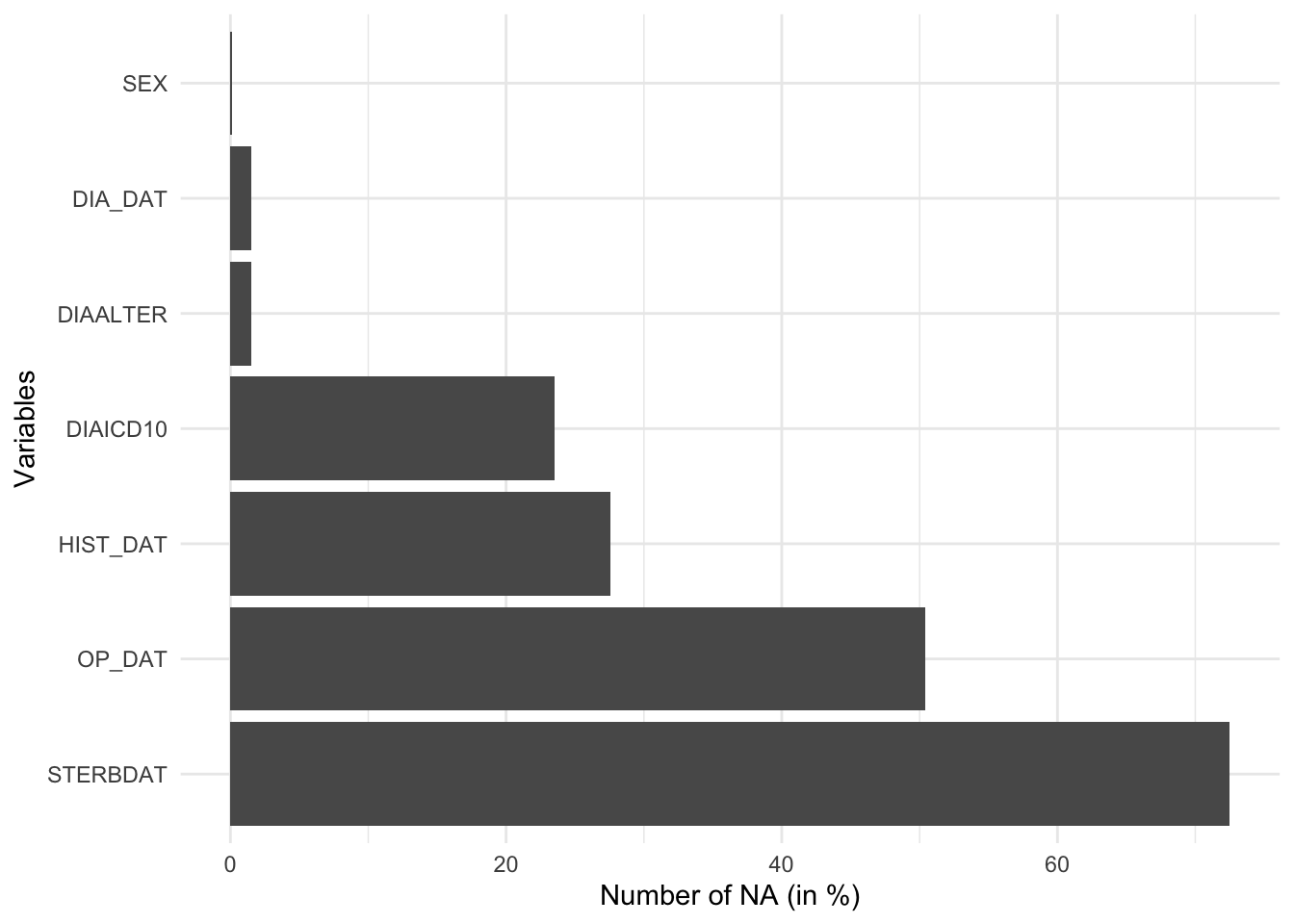
Nachfolgend nochmals unsere Auswertung zu fehlenden Werten, diesmal aber in einer druckreifen Grafik-Version, die dank der zugrunde liegenden Farb-Palette sogar für Menschen mit Farbsehstörung geeignet wäre. Parallel zur (im HTML-basierenden Format) angezeigten Grafik könnte über einen Einzeiler dieselbe Grafik auch zur weiteren Verwendung in verschiedenen Formaten wie PDF, JPEG, TIFF, PNG, BMP, SVG oder PDF abgespeichert werden - wie gesagt, druckfertig und in gewünschter Auflösung und Größe.
Diese Grafik erforderte wenige zusätzliche Festlegungen, die ohne Aufwand unter Verwendung des Bibliothekspaketes “ggplot2” 3 zu bewerkstelligen sind. Neben der Farbe fällt die geänderte, besser interpretierbare Skalierung der x-Achse (bis 100 %) auf 4.
Footnotes
R unterliegt GNU Lizenzen↩︎
ggplot2 ist ein sehr mächtiges Paket aus dem Tidyverse insbesondere zur grafischen Aufbereitung statistischer Daten.↩︎
Unter Verwendung eines Vorschlags von Jens Laufer↩︎